Identifier les virus qui combattent nos infections
La phagothérapie veut utiliser des virus pour tuer des bactéries – une alternative prometteuse aux antibiotiques, dont l’effectivité baisse face aux bactéries résistantes. Un nouvel algorithme permet de présélectionner les bons virus.
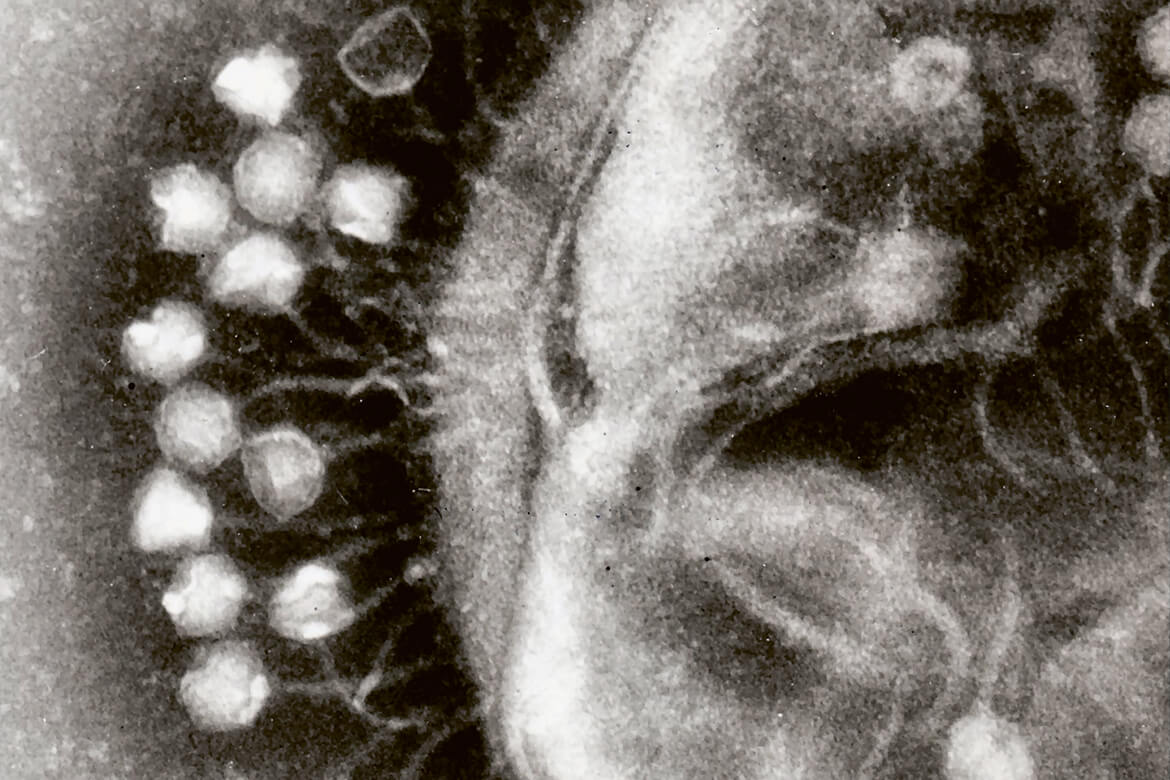
Des virus s’attachent à la paroi d’une bactérie avant de la détruire. | Image: Wikimedia Commons/Dr Graham Beards
Les phages sont très spécifiques et doivent être sélectionnés pour atteindre leur cible. Pour l’instant, cette étape se fait à l’aide de fastidieux tests menés en laboratoire. Afin d’accélérer ce triage, des scientifiques ont développé des modèles informatiques capables de prédire rapidement et à moindre coût les interactions phages-bactéries, basés sur l’analyse de leurs génomes. L’étude a été réalisée par l’Institut suisse de bioinformatique (ISB), la Haute école d’ingénierie et de gestion du canton de Vaud, l’Université de Lausanne et l’Hôpital universitaire de Berne.
Ces modèles prédictifs se basent sur les techniques d’apprentissage automatique supervisées, une approche statistique qui permet à un algorithme d’apprendre à partir de données. «Nous avons collecté des informations sur plus de mille interactions entre phages et bactéries, connues avec leurs génomes complets», indique Carlos Peña de l’ISB, qui a mené l’étude. Les scientifiques ont ensuite extrait de ces génomes des indicateurs donnant les caractéristiques propres des phages et des bactéries, comme la structure de certaines protéines et si elles se lient entre elles. Ces données ont ensuite été utilisée pour l’entraînement d’un algorithme, qui arrive à pronostiquer correctement environ 90% des interactions. «Bien sûr, les prédictions doivent toujours être testées en laboratoire, note le chercheur. Mais on pourra le faire de manière bien plus ciblée, ce qui permet un gain de temps considérable.»
D. M. C. Leite et al.: Computational prediction of inter-species relationships through omics data analysis and machine learning. BMC Bioinformatics (2018)